BioRecords
Über BioRecords
BioRecords ist eine integrierte Platform zur kollaborativen Sammlungsverwaltung
Geschichte
BioRecords wurde im Zuge des Projekts "Rote Liste der Wildbienen Österreichs" in Kooperation mit dem NHMW unter der Leitung von Mag. Esther Ockermüller (Büro für Entomologie und Naturschutz) und Dr. Herbert Zettel (NHMW) entwickelt.
In Ermangelung einer modernen Online-Multiuser-Datenbank, die die dezentrale Eingabe von Belegdaten schnell und effizient ermöglicht, wurde BioRecords entwicklet.
In Ermangelung einer modernen Online-Multiuser-Datenbank, die die dezentrale Eingabe von Belegdaten schnell und effizient ermöglicht, wurde BioRecords entwicklet.
Besonderheiten
Individuelle IDs sorgen für die Authentizität der Datensätze. Die flexible Benutzerverwaltung und die Unterteilung der Belege in Datensets ermöglicht optimale die Trennung der Belegdaten und die Gestaltung der Zugriffsrechte. Das Stichwort-System (engl. Tags) ermöglicht darüberhinaus alle möglichen Datensätze induviduell zu markieren und somit alle Entitäten sehr flexibel zu strukturieren.
Das interne Kommunikationssystem ermöglicht Kommentare direkt bei den Belegen, Orten, Personen, etc. zu hinterlegen. So können die Bearbeiter direkt am Datensatz Hinweise hinterlegen oder Probleme mit den anderen Bearbeitern besprechen. Da die Benutzer oft nicht persönlich untereinander bekannt sind bzw. nicht am selben Ort arbeiten - kann jeder Benutzer ein Bild von sich hochladen, das bei den Kommentaren angezeigt wird. Das macht die Kommunikation natürlicher und intuitiver.
Neben der obligaten Strukturierung der Arten in taxonomische Ebenen kann zu jeder Art die bekannten Verbreitungsgebiete, aus der Literatur, erfasst werden. Durch diese Daten werden die Bearbeiter über Neufunde informiert. So können die Belege unmittelbar bei der Eingabe ggfs. separiert bzw. gesondert etikettiert werden und so für zukünftige Publikationen bereitgestellt werden.
Alle Arten können mit Bilder versehen werden. Dadurch können auch ungeübte Bearbeiter schon bei der Eingabe die visuelle Übereinstimmung der Belege mit den selektierten Taxonen überprüfen.
Auch ist es möglich den Personen, neben den üblichen Stammdaten, Bestimmungsqualifikationen, in Bezug auf die vorhandenen taxonomischen Ebenen zuzuweisen, die Qualität der Determination kann so automatisch überprüft und den Bearbeiter - schon bei der Eingabe - mitgeteilt werden, ob (va. bei kritischen Arten) der Determination vertraut werden kann oder ob der Beleg separiert und erneut von fachkundiger Seite überprüft werden sollte.
Darüberhinaus werden Qualitätschecks bei jeder Beleg-Eingabe automatisch gemacht, sodass die Verwendbarkeit der Daten zur Gefährdungseinschätzung der Arten für die "Rote Liste" zu jeder Zeit ersichtlich ist.
UUIDs (Universelle einzigartige Identifikationsnummern) bei den Stammdaten und den Belegdaten ermöglichen einen möglichst fiktionsfreien Datenaustausch zwischen den Datenbanken bzw. Accounts.
Der Import und Export aller Felder und Daten gewährleisten die Unabhängikeit jedes Benutzers. Auch selektive Exporte sind möglich.
Mit dem integrierten Etikettensystem können Fund-, Determinations- und Individuelle-Nummern-Etiketten erstellt werden. Die effiziente Erstellung von Etiketten und die richtige Organisation bei der Eingabe ist wesentlich für schnelle die Eingabe von Beleg-Daten. Mit der Option QR-Codes auf den Etiketten platzieren zu können, werden bestimmte Informationen (z.B.: die Individuelle Nummer) maschinenlesbar gemacht.
Automatisch werden im Hintergrund diverse Auswertungen erstellt und an den verschiedenen Stellen visualisiert. So bekommt jeder Benutzer schon beim Einstieg (Dashboard) diverse Statistiken präsentiert. Z.B. wieviele Belege in letzter Zeit von ihm bzw. vom gesamten Team erstellt wurden oder ob neue Kommentare zu den Daten, die ihn betreffen, verfügbar sind.
Das interne Kommunikationssystem ermöglicht Kommentare direkt bei den Belegen, Orten, Personen, etc. zu hinterlegen. So können die Bearbeiter direkt am Datensatz Hinweise hinterlegen oder Probleme mit den anderen Bearbeitern besprechen. Da die Benutzer oft nicht persönlich untereinander bekannt sind bzw. nicht am selben Ort arbeiten - kann jeder Benutzer ein Bild von sich hochladen, das bei den Kommentaren angezeigt wird. Das macht die Kommunikation natürlicher und intuitiver.
Neben der obligaten Strukturierung der Arten in taxonomische Ebenen kann zu jeder Art die bekannten Verbreitungsgebiete, aus der Literatur, erfasst werden. Durch diese Daten werden die Bearbeiter über Neufunde informiert. So können die Belege unmittelbar bei der Eingabe ggfs. separiert bzw. gesondert etikettiert werden und so für zukünftige Publikationen bereitgestellt werden.
Alle Arten können mit Bilder versehen werden. Dadurch können auch ungeübte Bearbeiter schon bei der Eingabe die visuelle Übereinstimmung der Belege mit den selektierten Taxonen überprüfen.
Auch ist es möglich den Personen, neben den üblichen Stammdaten, Bestimmungsqualifikationen, in Bezug auf die vorhandenen taxonomischen Ebenen zuzuweisen, die Qualität der Determination kann so automatisch überprüft und den Bearbeiter - schon bei der Eingabe - mitgeteilt werden, ob (va. bei kritischen Arten) der Determination vertraut werden kann oder ob der Beleg separiert und erneut von fachkundiger Seite überprüft werden sollte.
Darüberhinaus werden Qualitätschecks bei jeder Beleg-Eingabe automatisch gemacht, sodass die Verwendbarkeit der Daten zur Gefährdungseinschätzung der Arten für die "Rote Liste" zu jeder Zeit ersichtlich ist.
UUIDs (Universelle einzigartige Identifikationsnummern) bei den Stammdaten und den Belegdaten ermöglichen einen möglichst fiktionsfreien Datenaustausch zwischen den Datenbanken bzw. Accounts.
Der Import und Export aller Felder und Daten gewährleisten die Unabhängikeit jedes Benutzers. Auch selektive Exporte sind möglich.
Mit dem integrierten Etikettensystem können Fund-, Determinations- und Individuelle-Nummern-Etiketten erstellt werden. Die effiziente Erstellung von Etiketten und die richtige Organisation bei der Eingabe ist wesentlich für schnelle die Eingabe von Beleg-Daten. Mit der Option QR-Codes auf den Etiketten platzieren zu können, werden bestimmte Informationen (z.B.: die Individuelle Nummer) maschinenlesbar gemacht.
Automatisch werden im Hintergrund diverse Auswertungen erstellt und an den verschiedenen Stellen visualisiert. So bekommt jeder Benutzer schon beim Einstieg (Dashboard) diverse Statistiken präsentiert. Z.B. wieviele Belege in letzter Zeit von ihm bzw. vom gesamten Team erstellt wurden oder ob neue Kommentare zu den Daten, die ihn betreffen, verfügbar sind.
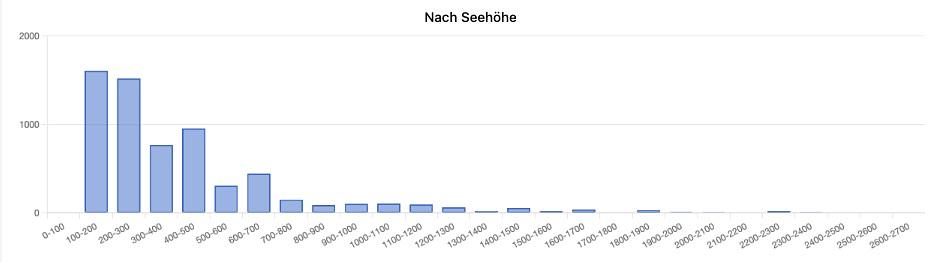
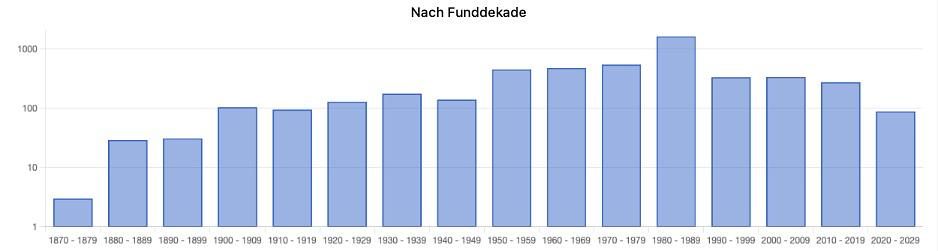
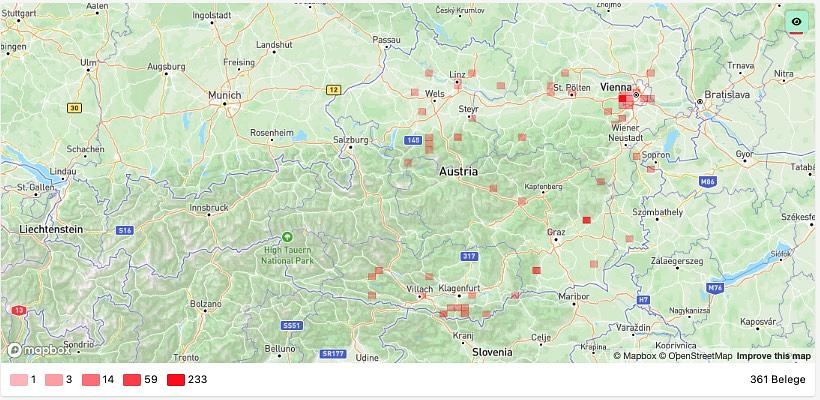
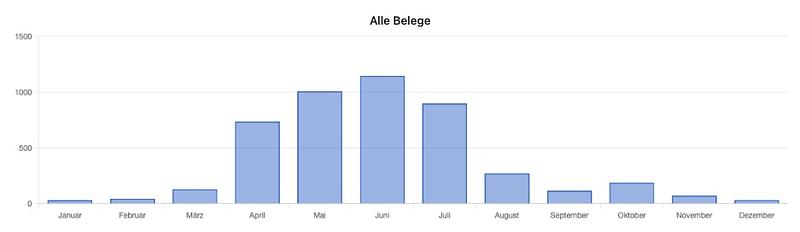
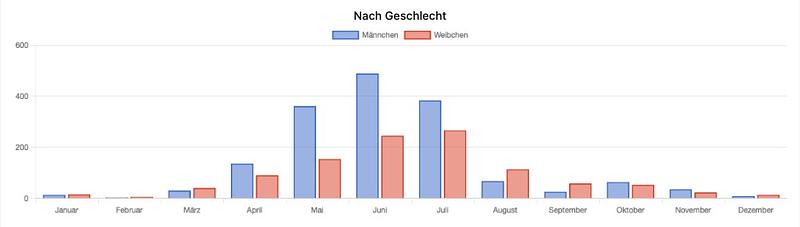
Das Auftreten der Arten im Jahresverlauf (Phänologie), ggfs. nach Geschlechtern getrennt, die Höhenverbreitung und die Aufteilung der Beleg-Daten über die Erhebungs-Dekaden werden in Diagrammen visualisiert und sind neben den obligatorischen Verbreitungskarten in Echtzeit (realtime) bei jeder Art ersichtlich.
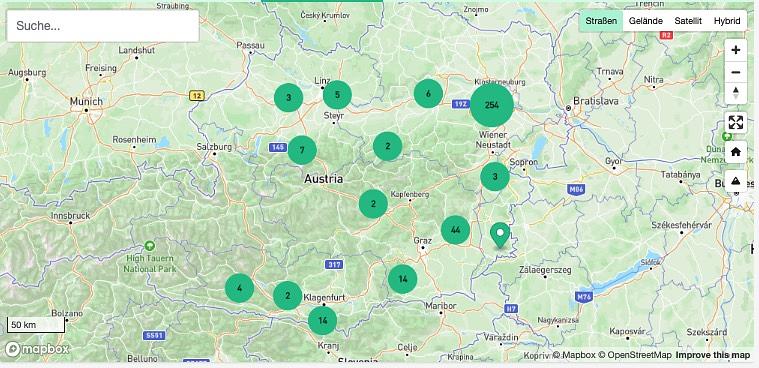
Alle Funde mit Koordinaten werden schon während er Eingabe mit den Polygonen der Regionen (Shapes) verschnitten. Dadurch findet eine automatische Zuordnung (Reverse Geocoding) zu den vorhandenen Region (Staaten, Bundesländer, Bezirke, etc.) statt. Es kann aber auch nachträglich jederzeit eine neue Region (zB.: ein Nationalpark) eingefügt werden. Diese Region wird ab dann in sämtliche Auswertungen mit einbezogen. On the fly können so Beleglisten, Artenlisten (Cheklisten), Verbreitungskarten sowie Listen für Neufunde für jede beliebige Region erzeugt werden.
Gerade bei importierten Daten schleichen sich immer wieder Fehleingaben ein, die eine größere Anzahl von Datensätzen betreffen. Ein typisches Problem ist auch die Vervielfachung von Stammdaten beim Import. Diese Probleme kann BioRecords mit dem Stapelverarbeitungs-Modul beheben. Hiermit können z.B. bei den Belegen über mehrere Datensätze hinweg Personen ergänzt oder korrigiert oder Tags ergänzt, korrigiert oder gelöscht werden. Auch können Datensätze aktiviert, deaktiviert oder sogar gelöscht werden. Ein mächtiges Tool, das allerdings vorsichtig eingesetzt werden sollte.
Darüberhinaus können aus allen markierten Datensätzen Rasterkarten erstellt werden, derzeit ist dieses Feature allerdings nur für Account-Admins verfügbar.